Pseudo-code non testé car ma pause déjeuner est presque terminée :
Vous avez un arbre à plusieurs racines, avec une racine principale choisie.
-
Pour chaque racine, créer un sous-graphe composé de tous les nœuds accessibles pour cette racine.
-
En commençant par la racine principale (racine a), calculez la distance/longueur du plus court chemin vers la racine pour tous les nœuds du sous-graphe A correspondant.
-
Trouvez tous les sous-graphes qui partagent au moins un nœud avec le sous-graphe principal, et sélectionnez le sous-graphe (sous-graphe B) qui a le nœud (nœud x) avec la plus petite distance à la racine principale.
-
Calculer la distance à la Racine b pour tous les noeuds du sous-graphe B. Ajouter la distance d(noeud x, Racine a). Soustrayez la distance d(noeud x, Racine b).
-
Créer l'union des sous-graphes A et B. Répéter les étapes 3 à 5 jusqu'à ce qu'il ne reste plus de racines.
-
Soustraire la distance maximale et inverser le signe de façon à ce que la racine principale ait la plus grande distance/valeur d'ordre.
Edit :
Mon pseudo-code fonctionne (*). Je blâme l'erreur de l'utilisateur ;-)
#!/usr/bin/env python
"""
https://stackoverflow.com/q/66584661/
"""
import numpy as np
import matplotlib.pyplot as plt
import networkx as nx
def hierarchical_layout(graph):
longest_path = nx.algorithms.dag.dag_longest_path(graph)
principal_root = longest_path[0]
roots = [node for node, degree in list(graph.in_degree) if degree==0]
subgraphs = {root : create_subgraph(graph, root) for root in roots}
# Starting with the principal root (root a), compute the
# longest path length to the root for all nodes in the
# corresponding subgraph A.
node_to_level = single_source_longest_dag_path_length(subgraphs[principal_root], principal_root)
explored = subgraphs[principal_root]
del subgraphs[principal_root]
while len(explored) < len(graph):
# Find all subgraphs that share at least one node with the
# principal subgraph, and select the subgraph (subgraph B) that
# has the node (node x) with the smallest distance to the
# principal root.
minimum_cost = np.inf
minimum_cost_node = None
minimum_cost_root = None
for root, subgraph in subgraphs.items():
for node in subgraph.nodes:
if node in node_to_level:
if node_to_level[node] < minimum_cost:
minimum_cost = node_to_level[node]
minimum_cost_node = node
minimum_cost_root = root
assert minimum_cost_node, "Could not find a connected subgraph."
# Compute the distance to the root b for all nodes in subgraph
# B. Add the distance d(node x, root a). Subtract the distance
# d(node x, root b).
path_lengths = [len(path) for path in nx.all_simple_paths(subgraphs[minimum_cost_root], minimum_cost_root, minimum_cost_node)]
offset = np.max(path_lengths) - 1
for node, distance in single_source_longest_dag_path_length(subgraphs[minimum_cost_root], minimum_cost_root).items():
if not node in node_to_level:
node_to_level[node] = distance + minimum_cost - offset
# Create the union of subgraph A and B.
explored = nx.compose(explored, subgraphs[minimum_cost_root])
del subgraphs[minimum_cost_root]
return node_to_level
def create_subgraph(G, node):
# https://stackoverflow.com/a/45678930/2912349
nodes = nx.single_source_shortest_path(G,node).keys()
return G.subgraph(nodes)
def single_source_longest_dag_path_length(graph, s):
# from AlaskaJoslin's comment to https://stackoverflow.com/a/60978007/2912349
dist = dict.fromkeys(graph.nodes, -float('inf'))
dist[s] = 0
topo_order = nx.topological_sort(graph)
for n in topo_order:
for s in graph.successors(n):
if dist[s] < dist[n] + 1:
dist[s] = dist[n] + 1
return dist
if __name__ == '__main__':
# edge_list = [
# ("n10", "n11"),
# ("n11", "n12"),
# ("n12", "n13"),
# ("n13", "n14"),
# ("n20", "n21"),
# ("n20", "n14"),
# ("n21", "n22"),
# ("n22", "n23"),
# ("n30", "n23"),
# ("n30", "n31"),
# ("n31", "n32"),
# ]
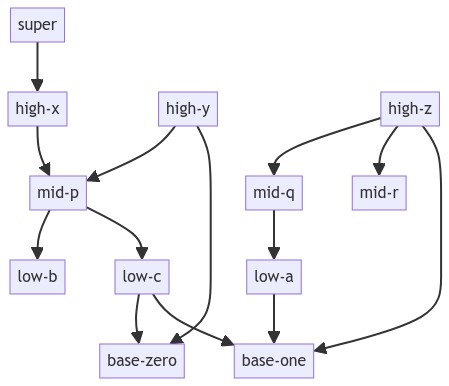
edge_list = [
("low-a", "base-one"),
("low-c", "base-zero"),
("low-c", "base-one"),
("mid-p", "low-b"),
("mid-p", "low-c"),
("mid-q", "low-a"),
("high-x", "mid-p"),
("high-y", "mid-p"),
("high-y", "base-zero"),
("high-z", "mid-q"),
("high-z", "mid-r"),
("high-z", "base-one"),
("super", "high-x"),
]
graph = nx.DiGraph()
graph.add_edges_from(edge_list)
node_to_level = hierarchical_layout(graph)
# reverse output format
distance_nodes_map = dict()
max_distance = np.max(list(node_to_level.values()))
for node, distance in node_to_level.items():
reversed_distance = max_distance - distance
if reversed_distance in distance_nodes_map:
distance_nodes_map[reversed_distance].add(node)
else:
distance_nodes_map[reversed_distance] = set([node])
# print(distance_nodes_map)
for ii, nodes in sorted(distance_nodes_map.items())[::-1]:
print(f"{ii} : {nodes}")
Rendement :
# 4 : {'super'}
# 3 : {'high-x', 'high-y', 'high-z'}
# 2 : {'mid-p', 'mid-r', 'mid-q'}
# 1 : {'low-a', 'low-b', 'low-c'}
# 0 : {'base-one', 'base-zero'}
(*) "soustraire la distance d(nœud x, Racine b)" impliquait naturellement la le plus long longueur du chemin entre le nœud x et la racine b. Évidemment.